蛋白质组研究的开展是生命科学研究进入后基因组时代的里程碑,是继基因组研究之后的又一“大数据科学”。蛋白质组是生物功能的执行者,在生命活动中发挥重要的作用。DNA结合蛋白预测研究是蛋白质组学和生物信息学的研究热点。深入研究DNA结合蛋白对于研究疾病发病机理、抗癌药物的研发以及药物靶标设计具有指导作用,对于解释和阐明生命活动的本质起着至关重要的作用。DNA结合蛋白不仅在维持基因信息的表达方面扮演着重要的角色,还在药物开发研究领域被用作抗生素、类固醇和抗癌药物的关键成分。随着高通量测序技术的开发和应用,新的蛋白质序列不断涌现,通过实验方法识别DNA结合蛋白已远远不能满足研究的需要,发展快速高效的人工智能方法对DNA结合蛋白的研究具有重要意义。

近日,青岛科技大学数理学院人工智能与生物医学大数据研究团队于彬副教授,在计算机科学一区TOP期刊Applied Soft Computing (IF=5.472) 上发表标题为“StackPDB: Predicting DNA-binding proteins based on XGB-RFE feature optimization and stacked ensemble classifier”的高水平论文,报道了构建预测DNA结合蛋白的人工智能算法模型—StackPDB。该模型表现出较强的泛化能力以及优异的计算稳定性。于彬副教授为论文的通讯作者,研究生张青梅为第一作者,青岛科技大学为第一完成单位。
本研究首次将梯度提升决策树和递归特征消除算法结合应用到DNA结合蛋白领域,对高维数据进行降维处理,降维后得到的低维子空间不仅可以保留有效信息,还可以剔除冗余和噪声。此外,本研究首次使用XGBoost、LightGBM和SVM组成的Stacking集成学习分类器,最严格的留一法检验结果表明,StackPDB预测模型表现了出色的DNA结合蛋白识别能力,给定蛋白质序列能够准确的识别出是否为DNA结合蛋白。通过与其它已报道的先进预测模型相比,StackPDB具有较快的运行速度以及较高的预测能力。本文提出的梯度提升决策树和递归特征消除算法相结合的策略,以及多种机器学习方法集成的方案,有望为新药研发和疾病治疗提供指导,也可用于生物信息学领域的其它重要问题研究。
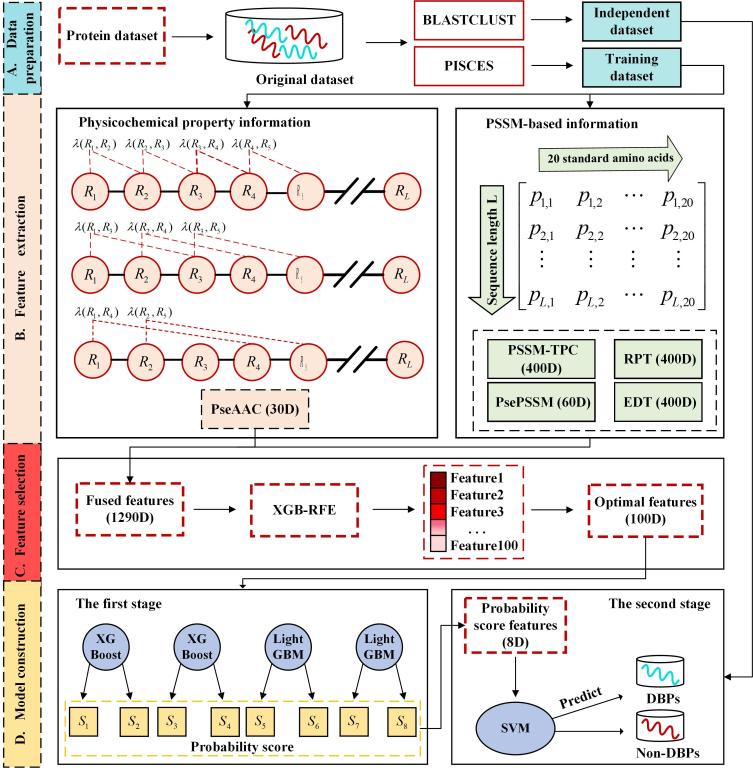
文章链接:https://doi.org/10.1016/j.asoc.2020.106921
此外,该课题组还在生物信息学研究领域的顶级期刊Bioinformatics取得两项关于蛋白质组学研究的重要成果,相关工作已发表论文:
Protein-protein interaction sites prediction by ensemble random forests with synthetic minority oversampling technique. Bioinformatics, 2019, 35(14): 2395-2402。数理学院本科生王筱颖为论文的第一作者,于彬副教授为通讯作者。
SubMito-XGBoost: predicting protein submitochondrial localization by fusing multiple feature information and eXtreme gradient boosting. Bioinformatics, 2020, 36(4): 1074-1081。于彬副教授为第一作者及通讯作者,硕士生邱文莹为第二作者。以上两篇论文均以青岛科技大学为第一完成单位。
Bioinformatics是牛津大学出版社 (Oxford Academic) 出版的JCR一区顶级期刊,2020年的影响因子为5.610,在SCI收录的227个“Mathematics”大类期刊中 “Statistics and Probability”小类排名第4。在SCI收录的146个“Mathematics”大类期刊中“Computational Mathematics”小类排名第5。
(撰稿:李磊 审核:王明辉)